基因测序(一、二、三代)
第一代测序:sanger测序
1、sanger测序原理
双脱氧链末端合成终止法
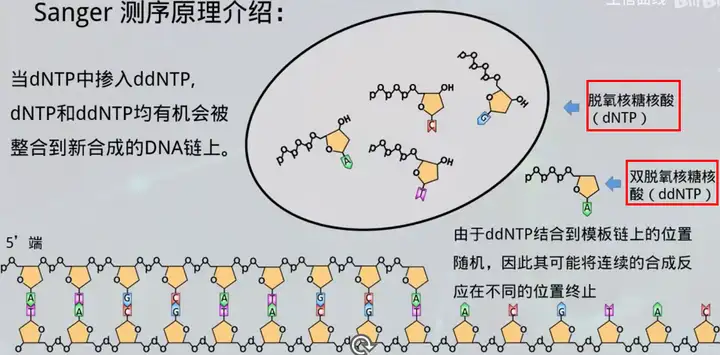
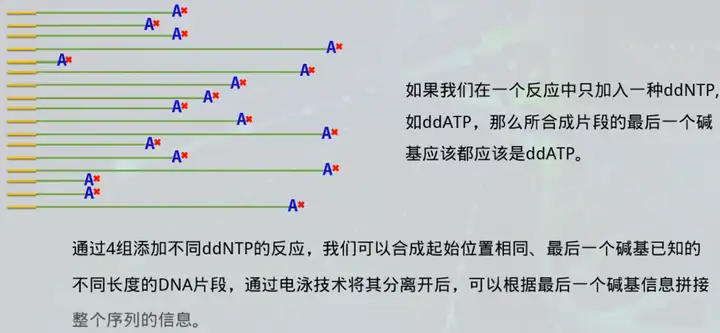
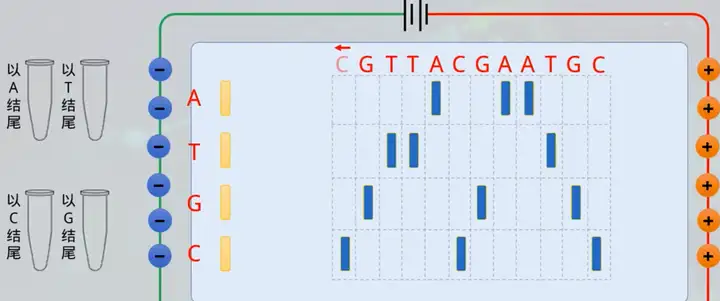
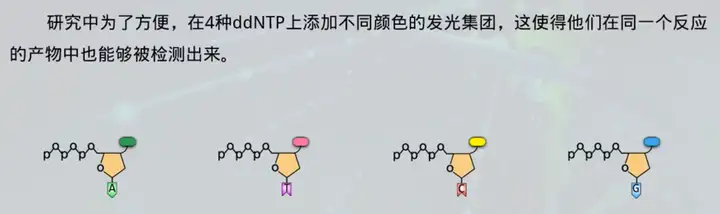
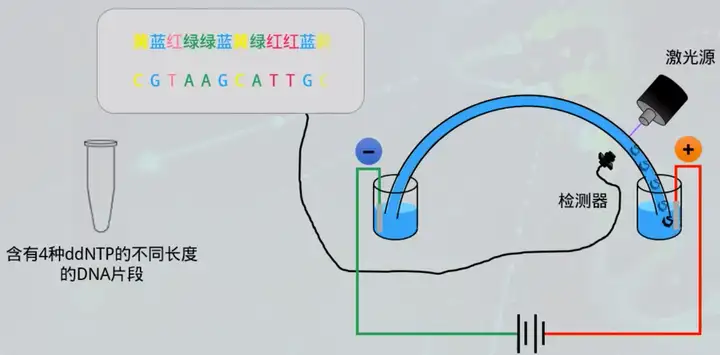
2、sanger测序的特点

第二代测序:illumina
1、什么是转录组
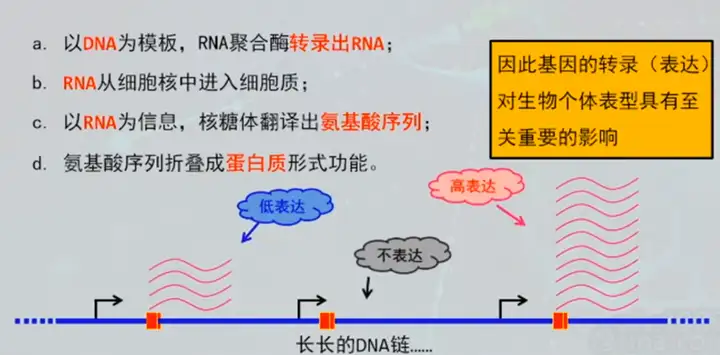
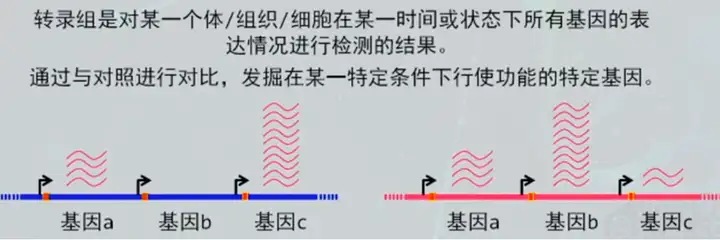


2、常见概念
NGS技术?
——next generation sequencing
最常见的就是illumina
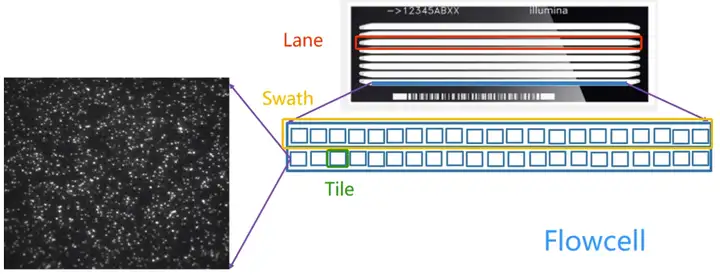
flowcell指illumina测序时发生反应的位置,一个flowcell含有8条lane(泳道,可以添加试剂、洗脱)
tile指每次测序荧光扫描的最小单位(一个lane中排列了无数个tile)
reads即测序结果,一条序列就是一条reads
bp base pair 碱基对——衡量序列长度
对于一条长500bp的序列,我们在两端各测150bp(采用的是双端测序)
中间留有的200bp是测不到的——junction
adapter 是测序中需要的特定序列(类似引物)
3、主要步骤
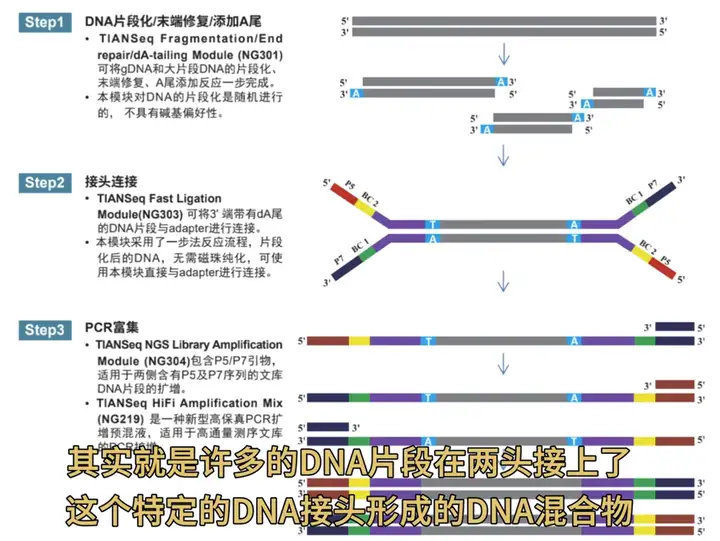
① Sample prep 即文库构建
illumina不适合太长的序列,比如X Ten就是用的双端150bp(一般测序时要先打断成300~500bp的片段,根据跑胶控制)
样本准备方法有很多种,不过所有制备方法都会在DNA片段末端加接头
==通过循环扩增的减少,额外的基序(motif)被引入==
打断后要考虑用酶补平不平整的末端——平末端
在3端加上一个特异的碱基A,通过碱基互补配对加上adapter(一部分是测序用的引物序列,一部分是建库扩增用的引物序列)
测序结合位点、标签(index/indices)、flowcell oligo(寡聚核苷酸引物互补区域)
PCR扩增,使得DNA样品符合上机要求
② Cluster generation
Flowcell 流动池(芯片)

簇生成是每个DNA片段被扩增的过程(增强信号)

桥式PCR扩增:
加入氢氧化钠碱溶液,发生解链,原始模板链被冲走(未与flowcell连接)
加入中性溶液,链发生折叠,与另一种引物发生配对
加入dNTP和酶,沿着第二类引物合成一条新链

再加入氢氧化钠……
形成簇
破坏特定引物上的基团以洗去反向链,留下正向链
如此循环会得到具有完全相同序列的簇——cluster
③ Sequencing
测序从第一个测序引物开始延伸,加入特殊处理过的A,T,C,G四种碱基(脱氧核糖3号位加入了叠氮基团而不是常规的羟基)
加入荧光标记的dNTP
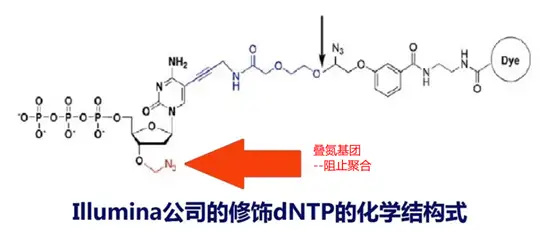
因此一个循环只能延长一个碱基——荧光修饰dNTP 可逆合成终止
此循环结束后用化学试剂去除叠氮基团和标记的荧光基因,暴露3’-OH,再加入dNTP和酶延长
边合成边测序
在大规模并行中,千万簇都被测序
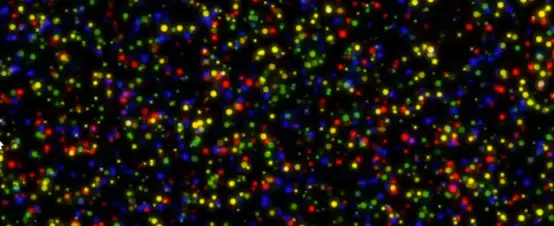
读段结束后,洗掉,检测index
加入index1测序引物,读6-8个碱基,可以知道片段来源于哪个样本
双端测序——有效长度增加一倍(index2)
Read2 测的是反向互补链
为什么Illumina测序会有长度限制?
经过长时间的PCR,比如一开始1个cluster中是100个完全一样的DNA链,但是经过1轮增加碱基,其中99个都加入了1个碱基,显示了红色,另外1个没有加入碱基,不显示颜色。这时候整体为红色,我们可以顺利得到结果。随后,在第2轮再加入碱基进行合成的时候,就变成了,之前没有加入的加入了1个碱基显示红色,剩下的99个显示绿色,这个时候就会出现杂信号。当测序长度不断延长,这个杂信号会越来越多,最后很有可能出现,50个红,50个绿色,这时候我们判断不出来到底是什么碱基被合成。
测序过程中,使用的碱基是特殊处理的,有一个非常大的荧光基团修饰。在使用DNA ploymerase的时候,酶的状态也会受到底物的影响,越来越差④ 数据分析

第三代测序:oxford nanopore


单分子测序的分辨率具有不可比拟的优势,而且没有PCR扩增步骤,就没有扩增引入的碱基错误。
该优势使其在特定序列的SNP检测,稀有突变及其频率测定中大显身手。
1、单分子荧光测序原理
代表性的技术为美国螺旋生物(Helicos)的SMS技术和美国太平洋生物(Pacific Bioscience)的SMRT技术。
脱氧核苷酸用荧光标记,显微镜可以实时记录荧光的强度变化。当荧光标记的脱氧核苷酸被掺入DNA链的时候,它的荧光就同时能在DNA链上探测到。当它与DNA链形成化学键的时候,它的荧光基团就被DNA聚合酶切除,荧光消失。这种荧光标记的脱氧核苷酸不会影响DNA聚合酶的活性,并且在荧光被切除之后,合成的DNA链和天然的DNA链完全一样。
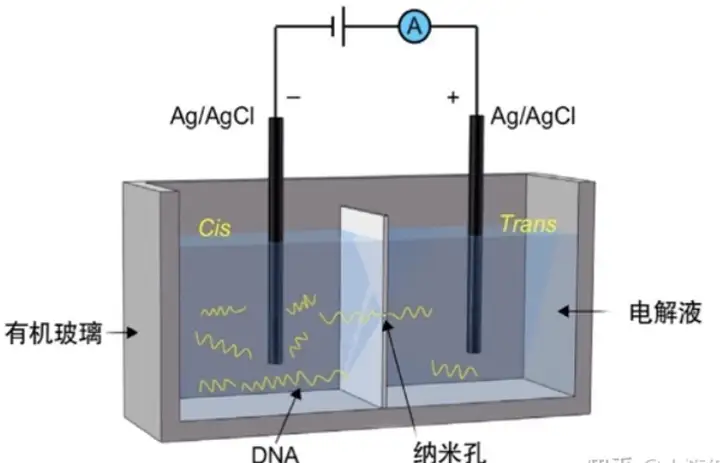
2、牛津纳米孔测序原理
新型纳米孔测序法(nanopore sequencing)是采用电泳技术,借助电泳驱动单个分子逐一通过纳米孔 来实现测序的。由于纳米孔的直径非常细小,仅允许单个核酸聚合物通过,而ATCG单个碱基的带电性质不一样,通过电信号的差异就能检测出通过的碱基类别,从而实现测序。
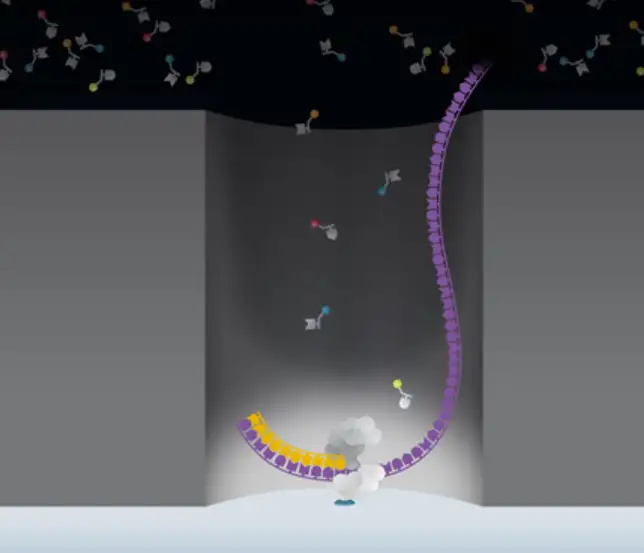
第一:因为在显微镜实时记录DNA链上的荧光的时候,DNA链周围的众多的荧光标记的脱氧核苷酸形成了非常强大的荧光背景。这种强大的荧光背景使单分子的荧光探测成为不可能。
Pacific Biosciences公司发明了一种直径只有几十纳米的纳米孔[zero-mode waveguides (ZMWs)],单分子的DNA聚合酶被固定在这个孔内。在这么小的孔内,DNA链周围的荧光标记的脱氧核苷酸有限,而且由于A,T,C,G这四种荧光标记的脱氧核苷酸非常快速地从外面进入到孔内又出去,它们形成了非常稳定的背景荧光信号。而当某一种荧光标记的脱氧核苷酸被掺入到DNA链时,这种特定颜色的荧光会持续一小段时间,直到新的化学键形成,荧光基团被DNA聚合酶切除为止。
第二:共聚焦显微镜实时地快速地对集成在板上的无数的纳米小孔同时进行记录。
技术特点
1、它实现了DNA聚合酶内在自身的反应速度,一秒可以测10个碱基,测序速度是化学法测序的2万倍。
2、它实现了DNA聚合酶内在自身的延续性,一个反应就可以测非常长的序列。二代测序可以测到上百个碱基,但是三代测序就可以测几千个碱基。
3、它的精度非常高,达到99.9999%。
4、直接测RNA的序列。既然DNA聚合酶能够实时观测,那么以RNA为模板复制DNA的逆转录酶也同样可以。RNA的直接测序,将大大降低体外逆转录产生的系统误差。
5、第二个是直接测甲基化的DNA序列。实际上DNA聚合酶复制A、T、C、G的速度是不一样的。正常的C或者甲基化的C为模板,DNA聚合酶停顿的时间不同。根据这个不同的时间,可以判断模板的C是否甲基化。本站所有文章、数据、图片均来自互联网,一切版权均归源网站或源作者所有。
如果侵犯了你的权益请来信告知我们删除。邮箱:dacesmiling@qq.com





